在这篇文章中,我们探索如何替使用编译后的WebAssembly替换缓慢的JavaScript计算来加速web应用。
如果你没有听说过,那这就是TL;DR(Too long; Don't read):WebAssembly是一个与JavaScript并行运行在浏览器的新语言。 是的,你没有听错,JavaScript不再是唯一一个可以在浏览器上运行的语言了。
但不仅仅是"不是JavaScript",它的区别因素是你可以将如C/C++/Rust(还有更多)等语言编译成WebAssembly,并在浏览器上运行它们。因为WebAssembly是静态类型,使用线性内存,以紧凑的二进制格式存储,所以它也非常快,并且最终允许我们以"接近本机"的速度运行代码,即以接近通过在命令行上运行二进制文件所获得的速度运行代码。利用现有工具和库在浏览器中使用的能力以及相关的加速潜力,是使WebAssembly对Web如此具有吸引力的两个原因。
到目前为止,WebAssembly以及被用于各种应用程序,从游戏(如:Doom 3)到将桌面应用程序一直到Web(如:Autocad和Figma)。它甚至被用于浏览器之外,例如作为一个高效和灵活的无服务计算(serverless computing)语言。
这篇文章是使用WebAssembly加速一个Web数据分析工具的实例学习。为了达到这个目的,我们将一个已经存在的使用C写的工具执行相同的计算,将它编译成WebAssembly,并使用它替代缓慢的JavaScript计算。
注意:这个将深入探讨一些高级主题,如编译C代码,但如果你没有这方面的经验,则无需担心。你仍然可以继续学习并了解WebAssembly可能实现的功能。
背景
我们将使用的Web应用程序是fastq.bio,这是一个交互式Web工具,它为科学家提供了DNA测序数据质量的快速预览,测序是我们读取DNA样本中"字母"(即核苷酸)的过程。
下面是应用程序的实际屏幕截图:
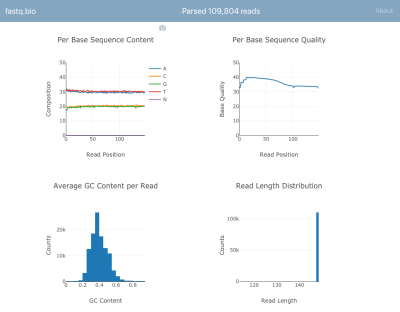
我们不会深入研究计算的细节,但简而言之,上面的图为科学家们提供了一种判断排序进行情况的判断,并被用来识别数据质量问题。
尽管有几十种命令行工具可用于生成此类质量控制报告,fastq.bio的目标是在不离开浏览器的情况加提供数据质量的交互式预览。这对于不熟悉命令行的科学家们尤其有用。
应用程序的输入是一个纯文本文件,有测序仪输出,其中包含DNA序列列表和DNA序列中每个核苷酸的质量分数。该文件的格式为"FASTQ",一次命名为fastq.bio。
如果你对FASTQ格式(对理解本文内容不是必要的)很好奇,查看FASTQ的Wikipedia页面。
Fastq.Bio:JavaScript实现
在 fastq.bio 的原始版本中,用户首先从计算机中选择一个FASTQ文件开始。对于文件对象,应用程序从随机字节位置开始读取一小部分数据(使用FileReader API)。在该数据块中,我们使用JavaScript执行基本的字符串操作并计算相关度量。一个这样的度量标准帮助我们追踪我们通常在DNA片段的每个位置上看到的A、C、G和T的数量。
一旦计算出该数据块的度量值,我们就可以使用Plotly.js绘制交互的结果,并移动到文件中的下一个块。以小块形式处理文件的原因仅仅是为了提高用户体验:一次处理整个文件会花费太长时间,因为FASTQ文件通常有数百GB。我们发现,块大小介于0.5M到1MB之间会使应用程序更加无缝 ,并更快递将信息返回给用户,但这个数字会因应用程序的详细信息和计算量的不同而有所不同。
我们最初的JavaScript实现的架构相当简单:
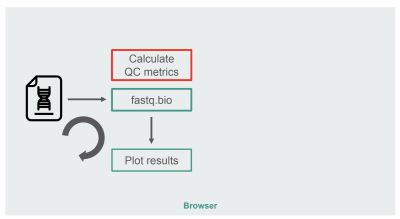
红色框是我们进行字符串操作生成度量的地方。该框是应用程序中计算量更大的部分,这自然使它成为使用WebAssembly进行运行时优化的好候选。
Fastq.Bio:WebAssembly实现
为了探索我们是否可以利用WebAssembly来加速我们的Web应用程序,我们搜索了一个县城的工具来计算FASTQ文件上的质量控制指标。具体地说,我们寻求了一种用C/C++/Rust编写的工具,使它适合移植到WebAssembly,并且已经被科学界验证和信任。
(未完全翻译,剩余内容请阅读原文)
